
Accélération des opportunités de découverte de nouveaux traitements contre la COVID-19
Aujourd'hui, il n'existe que quelques traitements approuvés dans le traitement de la COVID-19, mais même s'il faut parfois des décennies et des millions de dollars pour développer de nouveaux traitements, serait-il possible d'utiliser des médicaments existants pour élaborer de nouveaux traitements ? Notre tout dernier rapport CAS Insights présente la manière dont CAS Knowledge Graphs révèle de nouvelles connexions et informations permettant d'identifier les médicaments susceptibles d'être réutilisés.
La réutilisation de médicaments est essentielle pour accélérer le développement de traitements. Toutefois, il peut être très difficile d'assembler toutes les informations critiques et d'établir des connexions autour de nouvelles protéines, virus, cibles, voies et informations cliniques. Cela démontre la manière dont CAS Knowledge Graphs peut identifier les principaux candidats cliniques à réutiliser pour traiter la COVID-19.
Qu'est-ce qu'un graphique de connaissances ?
Un graphique de connaissances combine des données issues de sources disparates pour modéliser un domaine particulier. Il décrit les données dans des nœuds et des périphéries. Les nœuds représentent les différents points de données et les périphéries correspondent à la relation entre eux. L'image ci-dessous fournit un exemple simplifié de graphique de connaissances qui prédit les médicaments susceptibles d'inhiber l'inflammation vasculaire.
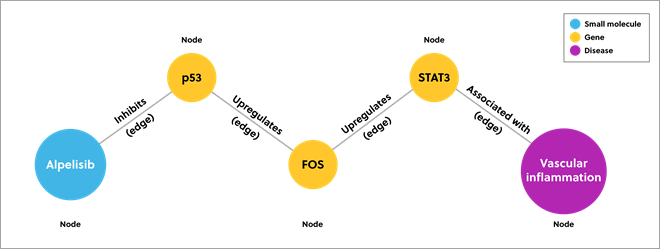
Figure 1. Exemple d'un graphique de connaissances présentant les relations entre les données à l'aide de nœuds et de périphéries
Les bases de données traditionnelles ne peuvent présenter que des relations directes (inhibiteurs directs du facteur de transcription STAT3), mais un graphique de connaissances affiche des relations plus approfondies entre les données. Dans cet exemple, le graphique de connaissances présente les inhibiteurs qui agissent plus loin sur le parcours.
Plongée dans la COVID-19 : découverte de médicaments à petites molécules
CAS Biomedical Knowledge Graph combine des données organisées par des scientifiques, issues de CAS Collection de contenusTM, et des données biomédicales publiquement disponibles.
Il contient des données de haute qualité provenant de plus de 6 millions de petites molécules, 24 000 maladies et 26 000 gènes humains et viraux. Un graphique de connaissances révèle des informations qui ne seraient pas accessibles par les méthodes de recherche traditionnelles.
Notre approche comprenait deux composants centraux pour découvrir des candidats médicaments potentiels contre la COVID-19 :
- Les scientifiques de CAS ont identifié 20 processus biologiques liés à la COVID-19. Ces processus comprenaient la coagulation sanguine, l'entrée virale et l'endocytose. Un nœud de maladie représentait le « choc cytokinique », un aspect important des formes graves de la pathologie de la COVID-19.
- Modifications de l'expression génique telles que constatées dans la littérature, plus précisément, les gènes nettement régulés à la hausse par l'infection au SARS-CoV-2. Ces modifications ont été utilisées pour identifier les processus biologiques pertinents et ceux qui étaient associés à 4 ou plus de ces gènes. Ces processus comprenaient la réponse inflammatoire, l'angiogenèse et la régulation négative de la transcription de l'ARN.
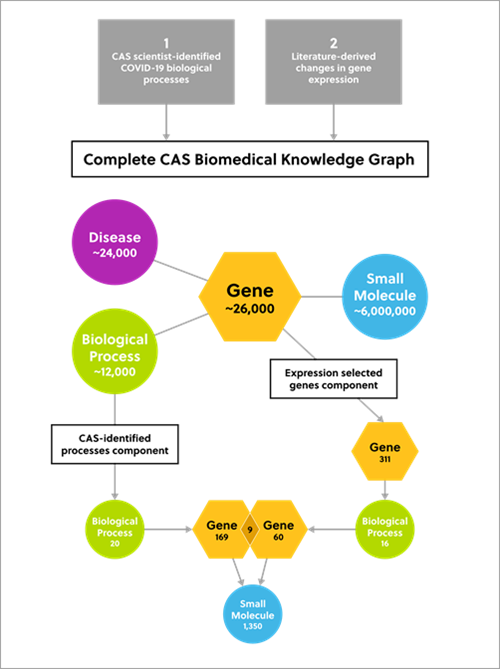
Figure 2. Schéma présentant l'approche à deux composants pour identifier les candidats médicaments potentiels à petites molécules pour le traitement de la COVID-19.
En utilisant le graphique de connaissances, nous avons identifié :
- Les éventuelles petites molécules qui inhibent ou activent les relations avec ces processus biologiques
- Les éventuelles petites molécules qui inhibaient les gènes régulés à la hausse
L'analyse a permis d'identifier 1 350 petites molécules qui pourraient être réutilisées en tant que traitement contre la COVID-19.
Évaluation des traitements potentiels de la COVID-19
Après avoir identifié les molécules potentielles, nous avons évalué le pouvoir de leurs relations et rehaussé les notes en conséquence. Pour ce faire, nous avons utilisé une nouvelle méthode algorithmique pour classer chaque molécule. L'équation a permis d'évaluer les relations entre les petites molécules et les interactions avec les gènes et les processus biologiques identifiés dans notre approche à deux composants.
Par exemple, un choc cytokinique était considéré comme une connexion importante. Nous avons ensuite évalué les relations entre les petites molécules et les interactions avec les gènes et les processus biologiques identifiés dans notre approche à deux composants. Les élévations de notes étaient accordées aux connexions importantes, comme le choc cytokinique, et aux petites molécules qui présentent une relation d'activation avec les gènes, compte tenu de la rareté de ces occurrences.
Nous avons ainsi réussi à établir un tableau de classement de toutes les petites molécules : les 50 premières sont présentées dans notre livre blanc. Dans la figure 2 ci-dessous, vous pouvez voir les 10 candidats médicaments les mieux notés en fonction des résultats. La taille du nœud correspond au nombre de connexions avec d'autres nœuds.
Figure 3. Schéma du réseau présentant les relations des 10 candidats médicaments les mieux notés établis à partir des résultats, où la taille des nœuds correspond au nombre de connexions à d'autres nœuds
Sur les 50 meilleurs médicaments identifiés dans notre tableau de classement, 11 sont actuellement en phase d'essai clinique pour le traitement de la COVID-19. Cela constitue une validation de nos résultats.
Notre graphique de connaissances biomédicales dévoile quatre catégories de médicaments qui ont précédemment été liées au SARS-CoV-2 ou aux mécanismes généraux d'infection virale. Les quatre catégories de médicaments sont les suivantes :
Inhibiteurs de kinase
Il s'agit de la plus importante catégorie de médicaments figurant dans nos résultats. Les kinases participent à la plupart des processus biologiques et leur activité est perturbée dans de nombreuses maladies. Les récepteurs à activité tyrosine kinase (RTK) participent à la pénétration de nombreux virus dans les cellules. Les inhibiteurs de kinase identifiés comprenaient ceux qui affectent les RTK, comme les récepteurs EGF, FGF, PDGF et ALK, ainsi que des non-récepteurs à activité tyrosine kinase, comme la tyrosine kinase de Bruton. Des inhibiteurs de sérine/thréonine kinases ciblant les récepteurs B-RAF, PKC, PIM et GSK-bêta-2 ont également été identifiés par notre graphique de connaissances.
Inhibiteurs de l'histone désacétylase (HDI)
Les HDI régulent l'expression génique en réduisant la désacétylation de l'histone. Les HDI réduisent à la fois l'expression de l'enzyme de conversion de l'angiotensine 2 (ACE2), le principal récepteur du SARS-CoV-2 à la surface des cellules, et celle de la glycosyltransférase ABO, l'enzyme qui participe à la régulation du groupe sanguin et qui est un facteur de risque connu pour la COVID-19. Les HDI régulent également plusieurs chimiokines et cytokines participant à la réponse immunitaire à la COVID-19. À ce titre, il est logique qu'ils soient inclus dans les résultats.
Agents régulateurs des microtubules
Les microtubules sont des filaments composés de sous-unités de tubuline. Des études ont démontré que les protéines du SARS-CoV-2 interagissent avec les microtubules ou avec les protéines associées aux microtubules. Nos résultats ont indiqué que les agents régulateurs des microtubules, comme le docétaxel, la colchicine et le mébendazole, pourraient être utiles pour perturber l'infection au SARS-CoV-2. La colchicine est déjà étudiée dans le cadre d'essais cliniques pour le traitement des patients atteints de la COVID-19.
Inhibiteurs de protéases
La plupart des inhibiteurs de protéases identifiés étaient des inhibiteurs du protéasome. Des études ont démontré que le système d'uniquitine-protéasome participe à la réplication virale et au choc cytokinique, y compris dans les maladies associées au coronavirus. Les inhibiteurs de protéases constituent un choix logique à explorer en relation avec la COVID-19. En fait, plusieurs inhibiteurs de ce type font déjà l'objet de recherches en tant que traitements contre la COVID-19. Certains figuraient dans nos résultats, notamment le bortézomib, le carfilzomib et la saxagliptine.
Le pouvoir des connexions
La méthodologie sous-jacente à notre graphique des connaissances renforce l'identification de médicaments potentiels contre la COVID-19 et sera précieuse pour la recherche portant sur d'autres maladies au-delà de la COVID-19, comme la maladie d'Alzheimer, la maladie de Parkinson, les maladies auto-immunes, le cancer et même des maladies rares. À la fois évolutifs et modulaires, nos graphiques de connaissances sont précieux dans tous les domaines de la science, y compris la chimie, la nutrition et les énergies renouvelables. Les opportunités sont multiples.
