
Depuis que l'Organisation Mondiale de la Santé a déclaré la COVID-19 comme une pandémie, les chercheurs ont énormément appris au sujet du SARS-CoV-2, le nouveau coronavirus à l'origine de cette maladie. Toutefois, malgré les multiples efforts et les lourds investissements réalisés, on n'a pas encore trouvé de traitements pour aider les patients atteints de COVID-19. Même si de nombreux candidats vaccins sont déjà en phase d'essai clinique dans le monde, même s'ils s'avèrent sûrs et efficaces, il faudra des mois, voire des années, pour fabriquer et distribuer les vaccins et les inoculer à l'ensemble de la population mondiale. Par conséquent, il est nécessaire de toute urgence d'identifier des traitements antiviraux efficaces, capable d'atténuer l'impact du virus sur les nombreuses autres personnes qui tomberont malades avant que cette pandémie ne soit maîtrisée.
Les scientifiques ont exploré différents moyens d'accélérer le processus de développement de médicaments pour répondre à ce besoin urgent, notamment en utilisant des approches informatiques pour identifier des médicaments déjà approuvés pour d'autres indications et susceptibles d'être efficaces dans le traitement de la COVID-19. Pour participer à cet effort, un groupe de scientifiques et d'experts technologiques de CAS a cherché à identifier des candidats médicaments potentiels pour traiter la COVID-19 avec des modèles d'apprentissage machine pour les cibles protéiniques prioritaires du SARS-CoV-2 en utilisant une méthodologie de relation quantitative structure-activité (QSAR). Ce travail, qui a permis d'identifier avec succès un certain nombre de médicaments qui commencent à présenter une efficacité clinique, notamment le Lopinavir et le Telmisartan, a été récemment publié dans ACS Omega.
Faire du neuf avec du vieux
Compte tenu du temps et des coûts substantiels nécessaires pour mettre un nouveau médicament sur le marché, la réutilisation de médicaments à petites molécules existants constitue une alternative attrayante, en particulier lorsque le besoin est très urgent. Outre l'accélération de la mise sur le marché des traitements, cette stratégie offre un certain nombre d'avantages par rapport au processus classique de développement de médicaments, notamment en réduisant le risque d'échec tardif dû à des effets secondaires indésirables.
La réutilisation de médicaments n'est pas un concept nouveau. Toutefois, son application à ce jour a été beaucoup plus opportuniste que systématique. Dans certains des exemples les plus efficaces de réutilisation de médicaments enregistrés jusqu'à présent, comme le Viagra et le Minoxidil, de nouvelles indications sont apparues suite à des effets secondaires inattendus signalés par des patients. Récemment, des approches plus systématiques de la réutilisation de médicaments ont été adoptées, notamment des méthodes informatiques comme la mise en correspondance des signatures, l'amarrage moléculaire, l'association génétique, la cartographie des voies et l'analyse clinique rétrospective. On espère qu'une approche informatique permettra aux chercheurs de corréler de manière fiable les traitements existants à petites molécules aux cibles médicamenteuses nouvellement identifiées, afin d'optimiser la valeur thérapeutique des portefeuilles existants.
Se rapprocher d'une cible
Les coronavirus sont une grande famille de virus dont on sait depuis longtemps qu'ils provoquent des maladies légères à modérées des voies respiratoires supérieures chez les humains et différentes espèces animales. Même s'il est rare que des coronavirus spécifiques à des animaux infectent et se propagent chez les humains, à ce jour, trois coronavirus se sont avérés capables de franchir cette barrière : le SARS-CoV-1, le MERS-CoV et le nouveau SARS-CoV-2. Tous les trois sont des betacoronavirus qui proviendraient des chauves-souris. Compte tenu des similitudes entre ces virus et de leur progression vers la contagion humaine, les anciennes recherches réalisées sur le SARS et le MERS constituent un bon point de départ pour chercher des cibles pour les médicaments contre le SARS-CoV-2. Parmi toutes les protéines du SARS-CoV-2, la protéase 3-C (chymotrypsine)-like (3CLpro) et l'ARN polymérase ARN-dépendante (RdRp) sont deux cibles protéiniques idéales pour la modélisation QSAR, en partie en raison des similitudes importantes qu'elles partagent avec des protéines identifiées dans le SARS-CoV et le MERS-CoV ainsi que dans d'autres coronavirus connus.
La 3CLpro est une protéase requise pour permettre au coronavirus de diviser les peptides de polyprotéines en protéines fonctionnelles non structurelles individuelles (NSP). Lorsqu'on compare les séquences d'acides aminés et les structures de protéines, la 3CLpro a fait preuve d'une haute conservation entre le SARS-CoV-2 et les autres coronavirus humains. Elle affiche un chevauchement d'identités de séquences de 96 % avec le SARS-CoV-1, de 87 % avec le MERS-CoV et de 90 % avec le CoV humain. En conséquence, les inhibiteurs de 3CLpro identifiés dans les recherches antérieures liées au coronavirus sont des inhibiteurs prometteurs pour la 3CLpro du SARS-CoV-2, et les données sur la relation structure-activité (SAR) associée sont précieuses pour entraîner des modèles d'apprentissage machine à la recherche de nouveaux inhibiteurs de la 3CLpro du SARS-CoV-2.
La RdRp est l'enzyme principale utilisée par les virus ARN pour répliquer les génomes viraux dans les cellules hôtes. Une étude structurelle et une analyse de séquence de la RdRp du SARS-CoV-2 ont révélé que cette enzyme est très similaire à la structure de la RdRp du SARS-CoV-1 et contient plusieurs résidus d'acides aminés clés qui sont conservés dans la plupart des RdRp virales, y compris le HCV. Heureusement, plusieurs RdRp virales ont été largement étudiées en tant qu'inhibiteurs des virus à ARN, en particulier dans la recherche liée au HCV. En conséquence, les inhibiteurs existants de la RdRp pour les virus ARN, comme le HCV, peuvent fournir des informations précieuses pour le développement de médicaments inhibiteurs de la RdRp du SARS-CoV-2.
Hiérarchisation des traitements existants avec l'apprentissage machine
Les modèles d'apprentissage machine sont de plus en plus utilisés pour faciliter la découverte de médicaments au cours des dernières années. Plus précisément, le QSAR est souvent l'une des premières étapes du processus moderne de découverte de médicaments. En termes simples, les QSAR sont des modèles mathématiques qui approchent des propriétés biologiques ou physicochimiques complexes de substances chimiques basées sur des mesures quantitatives de leurs structures moléculaires. Ces modèles mathématiques prédictifs sont utilisés pour passer en revue de grandes bases de données de structures chimiques afin de hiérarchiser les candidats médicaments potentiels les plus susceptibles d'être actifs contre les cibles identifiées. Cette approche suppose que l'activité d'une substance chimique est directement liée à sa structure et, par conséquent, que les molécules présentant des fonctionnalités structurelles similaires auront des propriétés physiques et/ou des effets biologiques similaires.
Dans cette étude, mes collègues et moi-même avons étroitement collaboré pour créer des modèles QSAR hautement prédictifs pour les cibles protéiniques 3CLpro et RdRp. L'équipe, qui comprenait des spécialistes de l'informatique et des chimistes, a organisé plus de 1 000 inhibiteurs présentant des données de structure-bioactivité comme molécules de formation pour les modèles. Nous avons collecté des données issues des études de bio-essais actuelles sur le SARS-CoV-2, ainsi que des études existantes portant sur le SARS-CoV-1, le MERS-CoV et d'autres virus apparentés dans la Collection de contenus CAS. En utilisant ces données, nous avons appliqué un certain nombre d'algorithmes d'apprentissage pour créer plusieurs dizaines de modèles QSAR en sélectionnant les modèles les plus performants parmi eux, l'un ciblant la 3CLpro et l'autre ciblant la RdRp.
Lisez l'article complet Modèles d'apprentissage machine QSAR et leurs applications pour identifier les composés ciblant la 3CLpro et la RdRp en tant que traitements potentiels de la COVID-19 et des infections virales apparentées pour voir tous les modèles testés et les candidats potentiels arrivés en tête.
Nous avons utilisé les deux modèles QSAR ainsi obtenus pour sélectionner un vaste ensemble de candidats médicaments potentiels, parmi lesquels 1 087 médicaments approuvés par la FDA, près de 50 000 substances issues du jeu de données Composés candidats antiviraux contre la COVID-19 de CAS et environ 113 000 substances présentant une activité pharmacologique identifiée ou un rôle thérapeutique indexé par CAS dans les documents concernant le SARS, le MERS ou la COVID-19 publiés depuis 2003. En modélisant l'activité d'inhibiteur de la protéase comme une fonction de la structure de la substance, nous avons identifié certains des candidats les plus prometteurs parmi les substances prévues comme devant être des inhibiteurs actifs de la 3CLpro et de la RdRp du coronavirus. En outre, un certain nombre de substances dont nos modèles prédisent qu'ils inhiberont la 3CLpro ou la RdRp du SARS-CoV-2 ont également une activité thérapeutique identifiée précédemment contre d'autres maladies apparues comme des facteurs de risque de développer des infections plus graves à la COVID-19. Par exemple, un candidat antiviral contre la COVID-19 qui possède aussi une activité connue contre les cardiopathies, comme le chlorhydrate de diltiazem (Cardizem) pourrait éventuellement fournir un double avantage dans certains cas.
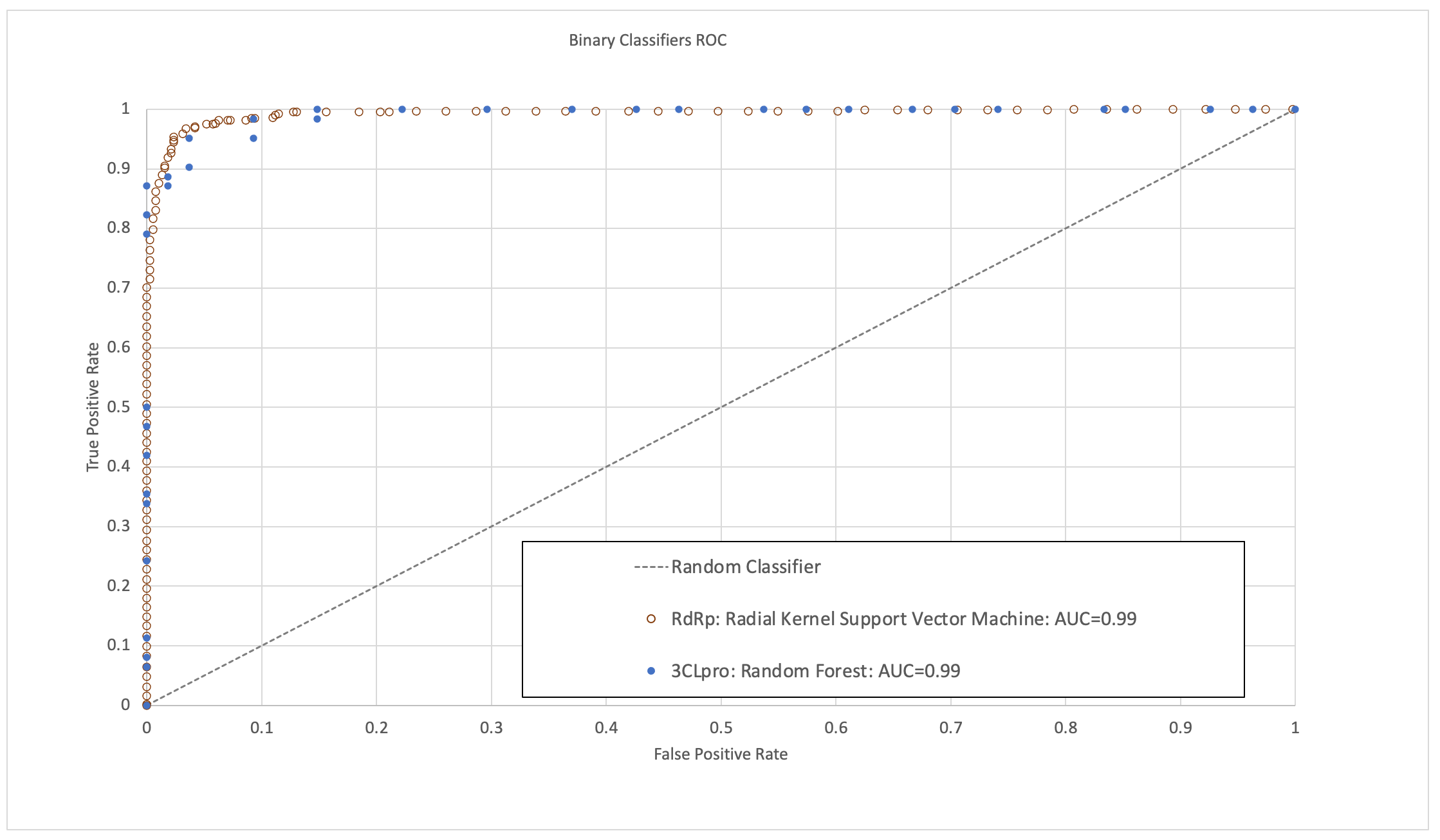
Les modèles ont été validés comme présentant une zone élevée sous la courbe de caractéristique opérationnelle du récepteur (ROC-AUC), la sensibilité, la spécificité et la précision (figure 1). Depuis la fin de cette recherche, certaines molécules dont on avait prédit qu'elles auraient une activité élevée dans ces modèles ont été validées par des études de bio-essais expérimentales et des essais cliniques publiés, ce qui apporte une nouvelle indication positive quant à leur aptitude prédictive.
Prendre une longueur d'avance sur la prochaine pandémie
Même si cette étude s'est concentrée sur l'identification de composés thérapeutiques potentiels à utiliser dans la crise actuelle de la COVID-19, il est probable que de nouvelles pandémies d'origine virale apparaîtront dans les années à venir. Il est donc urgent de commencer à se préparer dès maintenant aux futures épidémies en investissant de manière continue et en se concentrant sur la recherche d'agents antiviraux. Dans la mesure où différents types de virus peuvent provoquer des épidémies (par ex. coronavirus, virus de la grippe, virus Ebola, rétrovirus) et où les tests portant sur l'innocuité et l'efficacité de chaque nouveau médicament ou chaque nouvelle indication chez l'être humain demandent toujours beaucoup de temps, des agents antiviraux et des vaccins à large spectre seraient de la plus grande valeur.
Le développement continu de méthodes de découverte de médicaments basées sur l'informatique, comme les procédures d'apprentissage machine décrites ici, l'amarrage moléculaire et la sélection virtuelle, aura une importance majeure. L'augmentation régulière de la puissance de calcul informatique et le développement ininterrompu d'algorithmes de prédiction des amarrages et des structures et de techniques de détermination de la structure des cristaux des protéines faciliteront ces progrès. En outre, l'utilisation du criblage à haut débit, de technologies en « omique » et la réutilisation de médicaments déjà développés continueront à gagner en importance. Toutefois, ces nouvelles méthodes pilotées par la technologie ne remplaceront pas la recherche humaine en laboratoire, mais viendront la compléter en lui apportant une efficacité accrue. Nous espérons que cet effort, qui a associé l'organisation humaine des données et des modèles d'apprentissage machine pour identifier avec succès des candidats médicaments potentiels à petites molécules contre la COVID-19, soulignera la valeur d'une synergie entre humains et machines dans la découverte de médicaments, tout en contribuant aux efforts continus de la recherche d'antiviraux contre la COVID-19 et au-delà.
Dans le cadre de la communauté scientifique mondiale, CAS s'engage à exploiter tous ses actifs et ses capacités pour appuyer la lutte contre la COVID-19. Explorez nos ressources supplémentaires CAS COVID-19 en accès ouvert, y compris des informations scientifiques, des jeux de données en accès ouvert et des rapports spéciaux.
