La production de plastiques bon marché, durables et adaptables a explosé au cours des dernières décennies, car ils sont omniprésents dans notre vie ; toutefois, ce polymère autrefois désirable possède une face sombre. Il faut parfois des centaines d'années aux plastiques pour se dégrader et avec une production qui atteint des niveaux astronomiques (plus de 350 millions de tonnes par an dans le monde), la pollution par le plastique est l'une des préoccupations environnementales les plus pressantes auxquelles le monde est confronté.
Une quantité étourdissante de 150 millions de tonnes de plastique est envoyée dans les décharges ou libérée dans l'environnement chaque année et plus de 8 millions de tonnes sont transportées par les rivières et les fleuves vers les océans de la planète. La plus grande partie de ces plastiques ne se dégrade pas, mais se divise simplement en microparticules. Ces microplastiques bien documentés sont présents dans l'eau des océans, dans le corps des animaux marins et même dans les profondeurs du système gastroentérique des humains. La pollution par le plastique est l'un des problèmes environnementaux les plus critiques auxquels l'humanité est confrontée aujourd'hui et les chercheurs s'efforcent de trouver des réponses à ce problème préoccupant.
Dépolymérisation : résoudre le dilemme du recyclage des polymères
Les plastiques sont composés de polymères, de longues chaînes créées par des blocs de monomères répétitifs. La plus grande partie des plastiques couramment utilisés sont des thermoplastiques ou thermodurcissables. Les thermoplastiques, comme l'acrylique, le polyamide et le polyéthylène, s'assouplissent et se ramollissent à haute température et durcissent lorsqu'ils refroidissent. Cette propriété les rend relativement faciles à recycler, car ils peuvent être assouplis et remoulés pour former de nouveaux produits, même si la baisse de qualité limite cet avantage. Les plastiques thermodurcissants, comme le polyuréthane, la résine époxy et la résine mélamine, durcissent sous l'effet de la chaleur et sont pratiquement impossibles à recycler. Les difficultés du recyclage des thermoplastiques et des thermodurcissants signifie que tous les plastiques sont finalement destinés à contribuer à la contamination de l'environnement.
Pour parvenir à un véritable recyclage, avec une réutilisation ultérieure dans de nouveaux produits, les déchets plastiques doivent être ramenés à leurs monomères d'origine par un processus appelé dépolymérisation. Il s'agit d'un progrès technique essentiel nécessaire pour permettre une économie globale circulaire des matériaux. Dans les systèmes biologiques, la dépolymérisation complète en monomères peut être nécessaire à l'absorption et à la croissance des microbes.
Pour parvenir à la dépolymérisation, les scientifiques se sont tournés vers la nature, à la recherche d'enzymes microbiennes capables de décomposer les plastiques. En 2012, des chercheurs de l'université d'Osaka ont découvert une enzyme dans une pile de compost qui parvenait à décomposer l'un des plastiques les plus utilisés au monde : le polyéthylène téréphthalate (PET, numéro de Registre CAS 25038-59-9, formule (C10H8O4)n).
Cette enzyme, appelée cutinase de compost de branche à feuille (LLC), brise les liaisons entre les monomères de PET, mais est intolérante à la température de ramollissement du PET à 65 °C, qui la dénature après quelques jours de travail à cette température et limite son utilisation industrielle. Dans la mesure où la dépolymérisation ne peut intervenir que dans du plastique fondu, les enzymes doivent être stables à une température élevée.
Dépolymérisation du PET à double action provoquée par une bactérie terricole peu connue
Le PET est un thermoplastique et l'un des polyesters les plus largement utilisés. La production mondiale de PET est passée de 42 millions de tonnes en 2014 à 50 millions de tonnes en 2016 et devrait atteindre 87 millions de tonnes d'ici 2022.
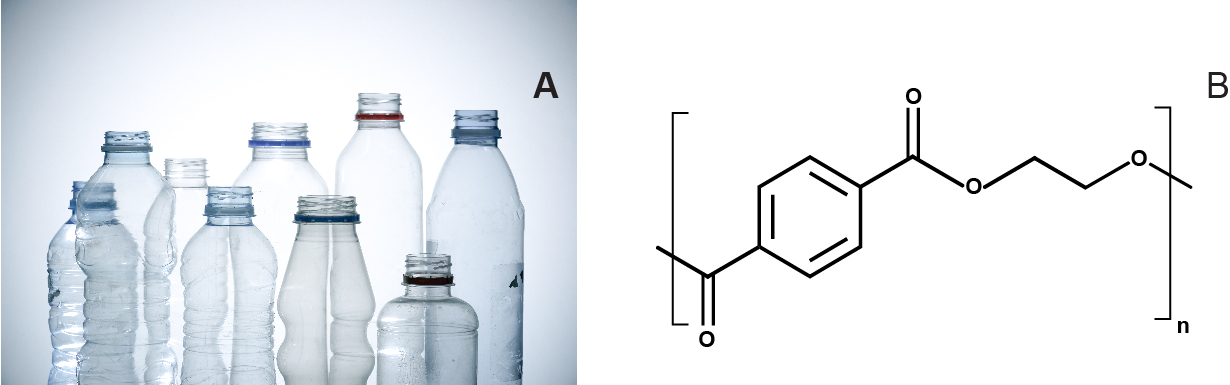
Ce polymère synthétique est fabriqué à base d'acide téréphtalique dérivée du pétrole (TPA) et d'éthylène glycol (EG). Le PET est un polymère polyvalent qui peut être rendu transparent, opaque ou blanc, selon la structure de ses cristaux et la taille des particules (fig. 1). Il est largement utilisé pour produire des fibres pour les vêtements et des conteneurs, y compris les bouteilles d'eau, et le PET non orienté peut être thermoformé (ou moulé) afin de fabriquer d'autres produits d'emballage, tels que des emballages-coques1. La recherche d'un moyen efficace de dépolymériser le PET sera un jalon majeur sur la voie d'un véritable recyclage du plastique et la protection de l'environnement que cela produira.
La biodégradation du PET a fait l'objet de nombreuses études, car les enzymes d'estérase (enzymes qui décomposent les esters en un acide et un alcool) sont abondantes dans la nature2. Les rapports sur la dégradation biologique du PET ou son utilisation pour soutenir la croissance microbienne sont cependant rares. Certains organismes du groupe des champignons filamenteux, Fusarium oxysporum et Fusarium solani, ont été cultivés sur un milieu minéral contenant des fils de PET3.
En 2016, Yoshida et al4 ont rapporté la découverte et la caractérisation de la souche de bactéries terricoles, Ideonella sakaiensis 201-F6, qui se développait autour de sédiments contaminés par du PET à proximité d'une usine de recyclage des plastiques au Japon. Cette bactérie en forme de bâtonnet, à gram négatif et aérobie, possède l'aptitude remarquable à utiliser le PET comme sa source majeure de carbone et d'énergie pour sa croissance.
I.sakaiensis utilise un système à deux enzymes pour dépolymériser le PET en le réduisant à ses éléments constitutifs, le TPA et l'EG, qui sont eux-mêmes catabolisés en une source de carbone et d'énergie. L'une des deux enzymes, la protéine ISF6_4831, hydrolyse et décompose les liaisons d'esters. Avec une préférence pour les esters aromatiques plutôt que les esters aliphatiques et une inclinaison particulière pour le PET, elle est désignée comme PET hydrolase (PETase). L'enzyme PETase dans I. sakaiensis est une hydrolase à sérine similaire à la cutinase qui attaque le polymère PET, libérant du bis(2-hydroxyéthyl) téréphthalate (BHET), du mono(2-hydroxyéthyl) téréphthalate (MHET) et du TPA. La PETase décompose encore le BHET en MHET et EG. La deuxième enzyme, la protéine ISF6_0224, hydrolase MHET (MHETase), hydrolyse davantage le MHET soluble pour produire du TPA et de l'EG (fig. 2). Les deux enzymes sont nécessaires, probablement de façon synergique, pour convertir le PET en ses deux monomères bénins pour l'environnement, le TPA et l'EG4, et ainsi recycler pleinement le PET.
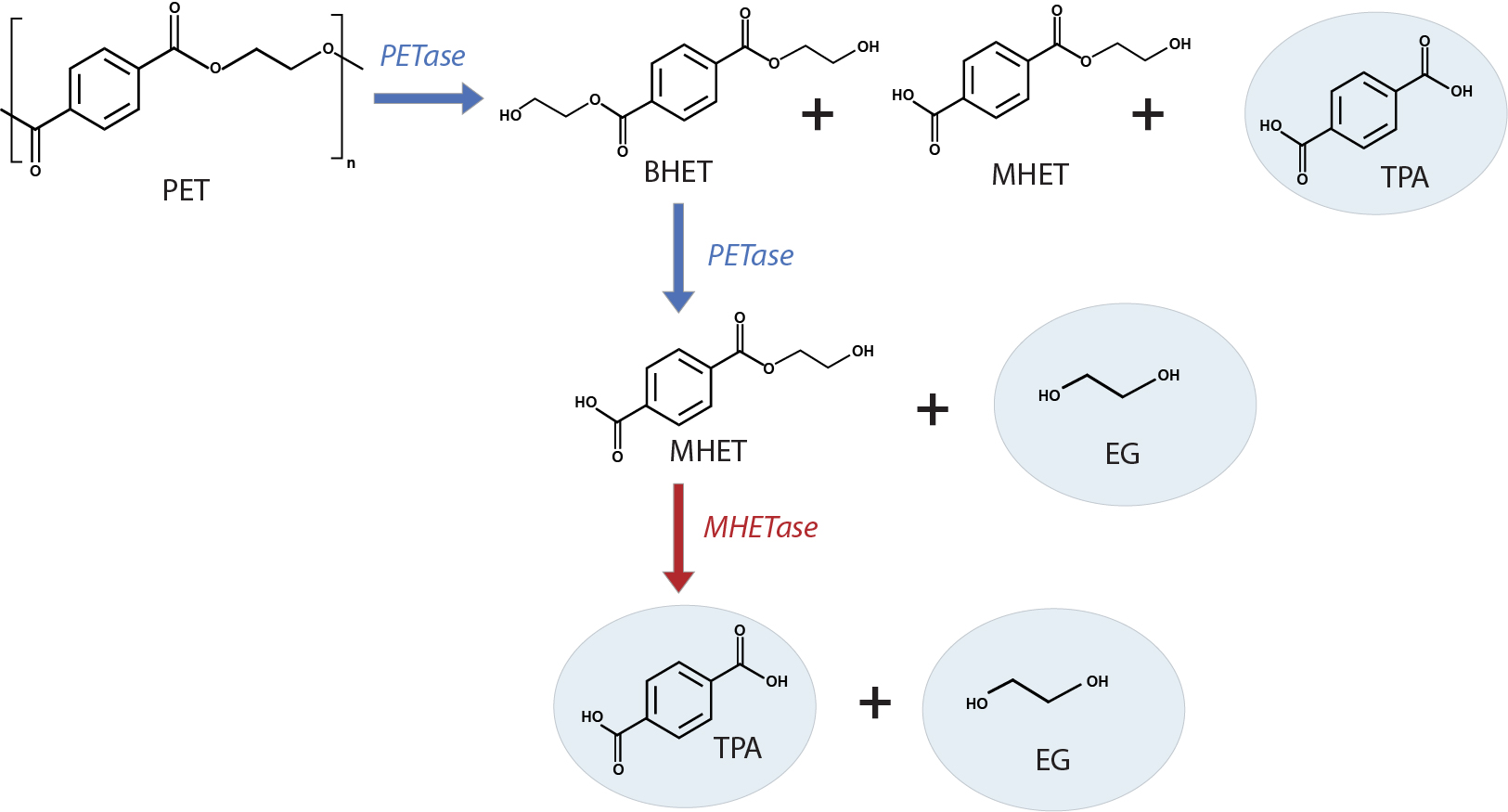
Fig. 2. Schéma de la dépolymérisation du PET : la PETase catalyse la dépolymérisation du PET en bis(2-hydroxyéthyl)-TPA (BHET), mono(2-hydroxyéthyl) téréphthalate (MHET) et en acide téréphthalique (TPA). La MHETase convertit le MHET en monomères TPA et éthylène glycol (EG).
Capacité de dégradation du PET du mutant de la PETase de grande taille
Les études de la séquence et de la structure de l'enzyme PETase mettent en évidence les similitudes avec les enzymes cutinases, produites par de nombreuses bactéries pour décomposer la cutine, un polymère cireux naturel qui fait partie de la cuticule protectrice de nombreuses plantes. L'analyse structurelle des cristaux et les tests biochimiques révèlent que la PETase présente dans I.sakaiensis 2 présente une architecture de site actif ouverte dans le site de liaison et fonctionne probablement selon le mécanisme de catalyse canonique de l'hydrolase à sérine5.
En fonction des modifications structurelles de la PETase et d'une fissure sur le site actif de la cutinase homologue, des variants de PETase ont été produits et testés pour la dégradation du PET, y compris un mutant présentant une double mutation distale du centre catalytique. On suppose que cette zone est capable d'amender des interactions importantes de liaison au substrat6. Ce double mutant, basé sur l'architecture de la cutinase, s'est avéré posséder des capacités avancées de dégradation du PET par rapport à la PETase de type sauvage6 et un brevet a maintenant été déposé7.
En réduisant la fissure de liaison par le biais de la mutation de deux résidus du site actif dans les cutinases, les chercheurs ont observé une dégradation améliorée du PET, ce qui suggère que la PETase ne présente pas la structure optimale pour la dégradation du PET cristallin, bien qu'elle évolue dans un environnement riche en PET. Il ne faut que quelques jours à l'enzyme mutante pour commencer à décomposer le plastique : c'est nettement plus rapide que les siècles nécessaires à cette décomposition dans les océans.
D'un double mutant à un cocktail à double enzyme
Lorsque l'enzyme MHETase est ajoutée à la réaction, le mélange d'enzymes décompose le PET deux fois plus rapidement que la PETase seule. La tendance à la dégradation observée dans la plage de charge de l'enzyme testée présente des niveaux accrus de monomères constitutifs à mesure que la concentration des deux enzymes augmente. Cela indique que les réactions sont limitées par l'enzyme, plutôt que par le substrat. L'analyse de la synergie indique également que le rythme de la dégradation augmente avec la charge de PETase et que la présence de MHETase, même à faible concentration par rapport à la PETase, améliore la dégradation totale. Les expériences actuelles n'indiquent pas un ratio optimal de PETase par rapport à la MHETase.
La création d'une super-enzyme triple la dégradation du PET
Dans d'autres expériences explorant les propriétés et la portée de la dégradation du PET, les chercheurs ont conçu une nouvelle super-enzyme en assemblant de la MHETase et de la PETase en une seule longue chaîne. Les protéines chimériques liant de manière covalente la terminaison C de la MHETase à la terminaison N de la PETase, en utilisant des liaisons glycine-sérine souples, ont été générées et évaluées pour la dégradation de PET amorphe (fig. 3). Lorsqu'on compare l'importance de la dégradation obtenue par différentes enzymes, les protéines chimériques dépassent à la fois la PETase et la MHETase, ainsi que le mélange d'enzymes non liées.
Il est intéressant de noter que la super-enzyme non seulement dépolymérise le PET, mais fonctionne aussi sur le PEF (polyéthylene furanoate), un bioplastique à base de sucre utilisé pour fabriquer les bouteilles de bière.
La déconstruction enzymatique de certains polymères naturels, comme la cellulose et la chitine, est obtenue dans la nature par des mélanges d'enzymes fonctionnant en synergie, secrétées par des microbes8. Ces systèmes microbiens naturels ont évolué au fil du temps pour dégrader de manière optimale de tels polymères. Il semble que certaines bactéries terricoles, comme I. sakaiensis, aient évolué de manière similaire pour utiliser un substrat de polyester avec un système à deux enzymes4,9. Contrairement à la dégradation naturelle, qui peut prendre des siècles, la super-enzyme est capable de convertir le PET en ses monomères en quelques jours seulement, même si cela reste trop lent pour être commercialement viable.
Recyclage infini avec dégradation du plastique
En convertissant le PET en ses monomères constitutifs d'origine, la super-enzyme permettrait aux plastiques d'être fabriqués et réutilisés à l'infini en réduisant la dépendance vis-à-vis des ressources fossiles. Et les avancées ne s'arrêtent pas là...
En 2020, une percée majeure a vu les scientifiques identifier une autre enzyme apte à dégrader le PET en 10 heures seulement10. La recherche a sélectionné une grande diversité de bactéries et d'enzymes comme candidats potentiels, notamment la cutinase de compost de branche à feuille (LLC), découverte en 2012. Des centaines d'enzymes mutantes de la PET hydrolase ont alors été produites en faisant varier les acides aminés sur le site de liaison et en améliorant la stabilité thermique. Les bactéries mutantes ont alors été passées en revue pour identifier celles qui décomposent efficacement le PET. Après avoir répété ce processus à maintes reprises, on a pu isoler une enzyme mutante qui est 10 000 fois plus efficace pour dégrader le PET que le LCC natif. Elle est également stable à 72 °C, proche de la température de fusion du PET. Cette découverte contribue de manière importante à la progression vers un recyclage à l'infini du PET et est déjà en phase pilote au niveau industriel10.
Nous ne voyons que la pointe de l'iceberg des possibilités que peuvent offrir ces microorganismes et leurs enzymes. La plupart des plastiques sont dérivés de combustibles fossiles, finis en termes de création et pourtant, omniprésents dans notre environnement. La pollution par le plastique restera un problème croissant si nous ne parvenons pas à trouver un moyen de former une économie circulaire. Dans quelques décennies seulement, nous ne serons plus en mesure de produire les articles en plastique dont nous dépendons si nous ne parvenons pas à trouver un moyen de recycler les déchets qui existent déjà. Le recyclage traditionnel n'est pas efficace ni durable et, si nous ne parvenons pas à réduire les plastiques à leurs monomères constitutifs à une échelle industrielle, nous ne pourrons pas espérer résoudre ce problème. Heureusement, avec l'aide de la nature, une certaine évolution compétente et une pincée d'ingéniosité scientifique, il reste un espoir de résoudre ce problème.
Vous aimeriez en savoir plus au sujet des innovations « vertes » ?
Dans la chasse aux nouvelles espèces en vue de dynamiser l'innovation, aucun lieu n'offre une biodiversité plus importante que les forêts tropicales du Brésil. Découvrez comment les données sont recueillies et exploitées aux fins d'un usage public et comment cela pourrait aider à protéger 15 à 20 % de la biodiversité de la terre.
(Lisez Organisation des données vitales pour libérer l'innovation offerte par la biodiversité du Brésil)
RÉFÉRENCES
(1) Pasbrig, E.; Claessens, P.; Walker, R. I.; Walker, R. Peelable cover film for pharmaceutical packaging, e.g. blister packs, comprises paper, aluminum foil or heat-resistant plastic, a layer of special plastic film, mesh or fabric, a layer of aluminum foil and a heat-sealing layer. EP1767347-A1; WO2007038488-A2; EP1928654-A2; AU2006294788-A1; US2008251411-A1; CN101316702-A; CA2623586-A1; JP2009509874-W; TW200727887-A; MX2008004201-A1; IN200801248-P2; ZA200802826-A; BR200616412-A2; WO2007038488-A3; EP1928654-A4.
(2) Han, X.; Liu, W. D.; Huang, J. W.; Ma, J. T.; Zheng, Y. Y.; Ko, T. P.; Xu, L. M.; Cheng, Y. S.; Chen, C. C.; Guo, R. T., Structural insight into catalytic mechanism of PET hydrolase. Nature Communications 2017, 8. DOI: 10.1038/s41467-017-02255-z
(3) Nimchua, T.; Eveleigh, D. E.; Sangwatanaroj, U.; Punnapayak, H., Screening of tropical fungi producing polyethylene terephthalate-hydrolyzing enzyme for fabric modification. J. Ind. Microbiol. Biotechnol. 2008, 35 (8), 843-850. DOI: 10.1007/s10295-008-0356-3
(4) Yoshida, S.; Hiraga, K.; Takehana, T.; Taniguchi, I.; Yamaji, H.; Maeda, Y.; Toyohara, K.; Miyamoto, K.; Kimura, Y.; Oda, K., A bacterium that degrades and assimilates poly(ethylene terephthalate). Science 2016, 351 (6278), 1196-1199. DOI: 10.1126/science.aad6359
(5) Rauwerdink, A.; Kazlauskas, R. J., How the Same Core Catalytic Machinery Catalyzes 17 Different Reactions: the Serine-Histidine-Aspartate Catalytic Triad of alpha/beta-Hydrolase Fold Enzymes. Acs Catalysis 2015, 5 (10), 6153-6176. DOI: 10.1021/acscatal.5b01539
(6) Austin, H. P.; Allen, M. D.; Donohoe, B. S.; Rorrer, N. A.; Kearns, F. L.; Silveira, R. L.; Pollard, B. C.; Dominick, G.; Duman, R.; El Omari, K.; Mykhaylyk, V.; Wagner, A.; Michener, W. E.; Amore, A.; Skaf, M. S.; Crowley, M. F.; Thorne, A. W.; Johnson, C. W.; Woodcock, H. L.; McGeehan, J. E.; Beckham, G. T., Characterization and engineering of a plastic-degrading aromatic polyesterase. Proc. Natl. Acad. Sci. U. S. A. 2018, 115 (19), E4350-E4357. DOI: 10.1073/pnas.1718804115
(7) Beckham, G. T.; Johnson, C. W.; Donohoe, B. S.; Rorrer, N.; McGeehan, J. E.; Austin, H. P.; Allen, M. D. New modified polyethylene terephthalate -digesting enzyme comprising amino acid mutation of an active site residue, is used to degrade a polymer e.g. polyester, aromatic polymer or semi-aromatic polymer and polyethylene terephthalate. WO2019168811-A1.
(8) Payne, C. M.; Knott, B. C.; Mayes, H. B.; Hansson, H.; Himmel, M. E.; Sandgren, M.; Stahlberg, J.; Beckham, G. T., Fungal Cellulases. Chem. Rev. 2015, 115 (3), 1308-1448. DOI: 10.1021/cr500351c
(9) Taniguchi, I.; Yoshida, S.; Hiraga, K.; Miyamoto, K.; Kimura, Y.; Oda, K., Biodegradation of PET: Current Status and Application Aspects. Acs Catalysis 2019, 9 (5), 4089-4105. DOI: 10.1021/acscatal.8b05171
(10) Tournier, V.; Topham, C. M.; Gilles, A.; David, B.; Folgoas, C.; Moya-Leclair, E.; Kamionka, E.; Desrousseaux, M. L.; Texier, H.; Gavalda, S.; Cot, M.; Guémard, E.; Dalibey, M.; Nomme, J.; Cioci, G.; Barbe, S.; Chateau, M.; André, I.; Duquesne, S.; Marty, A., An engineered PET depolymerase to break down and recycle plastic bottles. Nature 2020, 580 (7802), 216-219. DOI: 10.1038/s41586-020-2149-4