在过去几十年中,廉价、耐用和适应性强的塑料生产呈爆发式增长,塑料已经渗透到我们生活的方方面面,但这种曾经令人向往的聚合物有其有害的一面: 塑料的降解可能需要数百年的时间,而且其产量已达到天文数字(全球每年的产量超过 3.5 亿吨),塑料污染是当今世界面临的最紧迫的环境问题之一。
令人难以置信的是,每年有 1.5 亿吨塑料被填埋或释放到环境中,超过 800 万吨塑料随河水流入海洋。 其中大部分不会降解,而只是分解成微粒。 这些有据可查的微塑料存在于海水中、海洋动物体内,甚至在人类胃肠系统深处。 塑料污染是当今人类面临的最严峻的环境问题之一,研究人员一直在忙于寻找这个令人困惑的问题的答案。
解聚:解决聚合物回收难题
塑料由聚合物制成——由重复的单体砌块制成的长链。 大多数被广泛使用的塑料要么是热塑性塑料、要么是热固性塑料。 热塑性塑料,如丙烯酸、聚酰胺和聚乙烯,在高温下变得柔软、可塑,冷却后变硬。 该属性使热塑性塑料相对容易被回收利用,因为它们可以软化并被改造成新产品(尽管质量下降限制了效益)。 热固性塑料,如聚氨酯、环氧树脂和三聚氰胺树脂,加热时变硬,几乎不可能被回收利用。 回收热塑性和热固性塑料时面临的挑战意味着:所有塑料最终都注定会造成环境污染。
为了实现解聚,科学家们已经 放眼大自然, 寻找
能够分解塑料的微生物酶。 2012年,日本大阪大学的研究人员在堆肥中发现了一种酶,这种酶可以分解全球最常用的塑料之一:聚对苯二甲酸乙二醇酯(PET,CAS 登记号 25038-59-9,分子式 (C10H8O4)n)。
大阪大学研究人员发现的这种酶,被称为叶枝堆肥角质酶 (LLC),能够破坏PET单体之间的键,但对PET的65°C软化温度不耐受,所以在该温度下工作几天后,就会发生变性,从而限制了其工业实用性。 由于解聚只能在熔融的塑料中发生,所以酶必须在温度升高时保持稳定。
一种鲜为人知的土壤细菌对PET的双解聚作用
PET是一种热塑性塑料,也是使用最广泛的聚酯之一。 全球PET产量从2014年的 4200 万吨增长到2016年的5000万吨,到 2022 年将达到 8700 万吨。
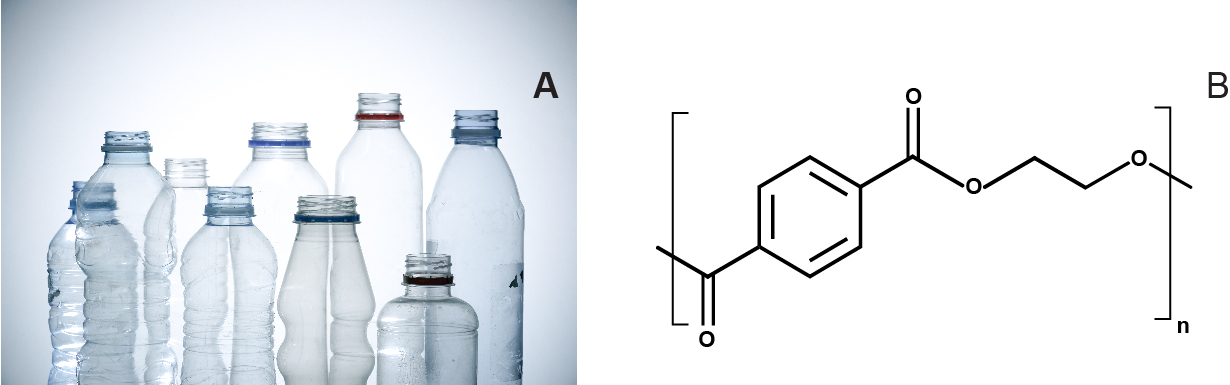
这种合成聚合物由石油衍生的对苯二甲酸 (TPA) 和乙二醇 (EG) 制成。 PET是一种多用途的聚合物,可根据晶体结构和粒径(图1)制成透明、不透明或白色的材料。 它被广泛用于生产服装纤维和容器,包括水瓶,无取向的PET可以被热成型(或模压)来制造其他包装产品,如泡罩包装1。 找到一种有效的解聚PET的方法将是实现真正的塑料回收和环境保护道路上的一个重要里程碑。
PET生物降解已被广泛研究,因为酯酶(将酯分解成酸和酒精的酶)在自然界中是丰富的2。 然而,关于PET的生物降解或利用PET支持微生物生长的报告并不常见。 一些来自丝状真菌群的生物,如尖孢镰刀菌和茄病镰刀菌,可以在含有 PET 纱线的矿物培养基上生长3。
2016 年,Yoshida 等人4报道了土壤细菌Ideonella sakaiensis 201-F6 的发发现及其特征,该菌株生长在日本一个塑料回收设施附近受PET污染的沉积物中。 这种革兰氏阴性、需氧、杆状的细菌具有非凡的能力,能够利用PET作为其生长所需要的主要碳源和能源。
I.sakaiensis采用双酶系统,将PET分解成其构建砌块TPA和EG,然后进一步分解为碳和能源。 两种酶之一的ISF6_4831蛋白质负责水解并断裂酯链。 ISF6_4831蛋白质更利于处理芳香酯,而不是脂肪族酯,对处理 PET尤其有用,所以它被指定为PET水解酶(PETase)。 I. sakaiensis中的 PETase酶是一种角质酶样丝氨酸水解酶,可攻击 PET聚合物,生成双(2-羟乙基)对苯二甲酸酯 (BHET)、单(2-羟乙基)对苯二甲酸酯 (MHET) 和 TPA。 PET水解酶进一步切开BHET,生成MHET和EG。 第二种酶,ISF6_0224蛋白,MHET水解酶(MHETase),进一步水解可溶性MHET生成TPA和EG(图2)。 这两种酶是必须的(可能通过协同作用),将PET酶转化为其两种对环境无害的单体——TPA和EG4,从而有可能完全回收PET。
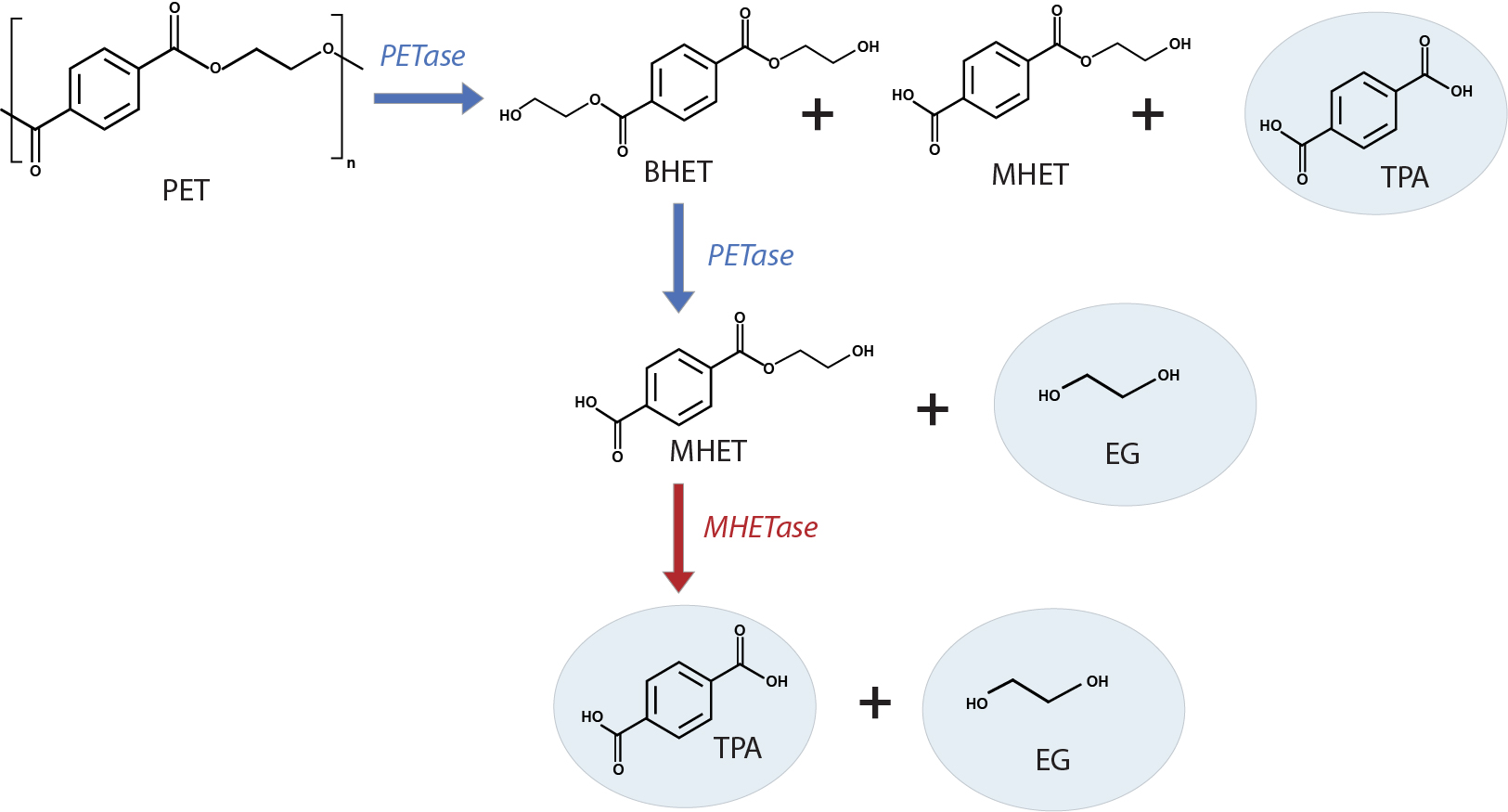
图 2. PET 解聚方法:PETase 可促进将 PET 解聚为双(2-羟乙基)-TPA (BHET)、单(2-羟乙基)对苯二甲酸酯 (MHET) 和对苯二甲酸 (TPA)。 MHETase 将 MHET 转化为单体 TPA 和乙二醇 (EG)。
PETase突变体的超强 PET降解能力
PETase的序列和结构研究强调了与角质酶的相似性,角质酶是由许多细菌进化而来分解角质的;是一种天然的蜡状聚合物,在许多植物中构成保护性角质层的一部分。 晶体结构分析和生化测试表明,I.sakaiensis 2 中的PETase在结合部位具有开放的活性位点结构,并且它可能遵循典型的丝氨酸水解酶催化机制进行工作5。
基于PETase的结构修饰和同源角质酶活性位点的裂解,现在已经生产出PETase变种,并用于测试PET降解,包括一个在催化中心远端的双突变变体。 假设该区域能够修正重要的底物结合相互作用6。 这种双突变体以角质酶结构为基础,表现出的 PET 降解能力比野生型 PETase6 更强,现已提交申请专利 7。
研究人员发现,通过角质酶中两个活性位点残基突变缩小结合裂口,PET 降解能力增强,这表明,尽管是在富含 PET 的环境中进化而成,PETase 并没有呈现最佳的结晶 PET 降解结构。 突变酶只需几天即可开始分解塑料,而在海洋中需要花上数百年,相比之下,这个速度快得惊人。
从双突变体到双酶鸡尾酒
当在反应中加入MHETase时,酶混合物分解PET的速度是PETase本身分解PET速度的两倍。 在试验酶载量范围内观察到的降解趋势发现,随着两种酶浓度的增加,组分单体的含量也在增加。 这表明反应受酶的限制而不是底物的限制。 协同效应分析还表明,降解率随着PETase负载量的增加而增加,并且因为MHETase的存在(即使在相对PETase较低的浓度下),也能提高总降解率。 目前的实验没有得出PETase和MHETase的最佳比例。
制造一种超级酶,使PET降解速度提高三倍
在进一步探索PET降解性质和范围的实验中,研究人员通过将MHETase和PETase拼接成一条长链,设计了一种新的超级酶。 利用柔性甘氨酸-丝氨酸连接子制备了MHETase的C末端与PETase的N末端共价连接的嵌合蛋白,用于无定形PET的降解(图3)。 在比较不同酶的降解能力时,嵌合蛋白的表现优于PETase和MHETase,未链接的酶混合物也是如此。
有趣的是,这种超级酶不仅能解聚PET,还能降解聚乙烯呋喃酸酯(PEF)。PEF是一种可用于啤酒瓶中的糖基生物塑料 。
一些天然聚合物(如纤维素和甲壳素)的酶解,是通过微生物分泌的协同作用酶的混合物在自然界中实现的8。 随着时间的推移,这些天然微生物系统已经进化到降解这些聚合物的最佳状态。 一些土壤细菌(例如大阪堺菌)似乎经历了类似进化,可以借助双酶系统来利用聚酯底物4,9。 自然降解可能需要几百年,而这种超级酶只需几天即可将 PET 恢复为单体,不过这离商业应用还很遥远。
通过塑料降解实现无限回收
使用超级酶将 PET 恢复为原始单体结构单元,塑料将可实现无限生产并重复使用,减少对化石资源的依赖。 还有更多突破……
2020 年,科学家取得了一项重大进展,他们发现了另一种酶,只需 10 小时即可降解 PET10。 研究筛选了许多种细菌和酶来寻找潜在候选物,包括 2012 年首次发现的叶枝堆肥角质酶 (LLC)。然后,通过改变结合位点的氨基酸、提高热稳定性,得到了数百种突变的 PET 水解酶。 然后对这些细菌突变体进行筛选,以找到有效的 PET 分解剂。 经过许多轮操作后,分离出了一种突变酶,其 PET 降解效率是天然 LCC 的 10,000 倍。 它在 72°C(接近 PET 的熔融温度)时也能保持稳定。 这一发现为实现 PET 无限回收做出了重大贡献,现已处于试生产阶段10。
这些微生物及其酶可以带来各种可能,我们发现的还只是冰山一角。 大多数塑料从化石燃料衍生而来,产量虽然有限,但在环境中无处不在。 如果找不到方法打造循环经济,塑料污染问题将日益严重。 除非找到方法回收现存废塑料,否则,短短几十年后,我们再也生产不了赖以生活的塑料制品。 传统回收要么效率低下,要么不可持续,除非大规模将塑料还原为单体成分,否则,解决这个问题的希望渺茫。 庆幸的是,大自然中有一些高明的进化,再加上一点科学创造,我们迎来了一线希望,这个问题或许可以解决。
有兴趣进一步了解“绿色”创新吗?
若要寻找新物种推动创新,可以去巴西的雨林,那里是世界上生物多样性最丰富的地方。 了解如何收集和挖掘数据供公众使用,以及这如何有助于保护地球上 15%-20% 的生物多样性。
参考文献
(1) Pasbrig, E.; Claessens, P.; Walker, R. I.; Walker, R. Peelable cover film for pharmaceutical packaging, e.g. blister packs, comprises paper, aluminum foil or heat-resistant plastic, a layer of special plastic film, mesh or fabric, a layer of aluminum foil and a heat-sealing layer. EP1767347-A1; WO2007038488-A2; EP1928654-A2; AU2006294788-A1; US2008251411-A1; CN101316702-A; CA2623586-A1; JP2009509874-W; TW200727887-A; MX2008004201-A1; IN200801248-P2; ZA200802826-A; BR200616412-A2; WO2007038488-A3; EP1928654-A4.
(2) Han, X.; Liu, W. D.; Huang, J. W.; Ma, J. T.; Zheng, Y. Y.; Ko, T. P.; Xu, L. M.; Cheng, Y. S.; Chen, C. C.; Guo, R. T., Structural insight into catalytic mechanism of PET hydrolase. Nature Communications 2017, 8. DOI: 10.1038/s41467-017-02255-z
(3) Nimchua, T.; Eveleigh, D. E.; Sangwatanaroj, U.; Punnapayak, H., Screening of tropical fungi producing polyethylene terephthalate-hydrolyzing enzyme for fabric modification. J. Ind. Microbiol. Biotechnol. 2008, 35 (8), 843-850. DOI: 10.1007/s10295-008-0356-3
(4) Yoshida, S.; Hiraga, K.; Takehana, T.; Taniguchi, I.; Yamaji, H.; Maeda, Y.; Toyohara, K.; Miyamoto, K.; Kimura, Y.; Oda, K., A bacterium that degrades and assimilates poly(ethylene terephthalate). Science 2016, 351 (6278), 1196-1199. DOI: 10.1126/science.aad6359
(5) Rauwerdink, A.; Kazlauskas, R. J., How the Same Core Catalytic Machinery Catalyzes 17 Different Reactions: the Serine-Histidine-Aspartate Catalytic Triad of alpha/beta-Hydrolase Fold Enzymes. Acs Catalysis 2015, 5 (10), 6153-6176. DOI: 10.1021/acscatal.5b01539
(6) Austin, H. P.; Allen, M. D.; Donohoe, B. S.; Rorrer, N. A.; Kearns, F. L.; Silveira, R. L.; Pollard, B. C.; Dominick, G.; Duman, R.; El Omari, K.; Mykhaylyk, V.; Wagner, A.; Michener, W. E.; Amore, A.; Skaf, M. S.; Crowley, M. F.; Thorne, A. W.; Johnson, C. W.; Woodcock, H. L.; McGeehan, J. E.; Beckham, G. T., Characterization and engineering of a plastic-degrading aromatic polyesterase. Proc. Natl. Acad. Sci. U. S. A. 2018, 115 (19), E4350-E4357. DOI: 10.1073/pnas.1718804115
(7) Beckham, G. T.; Johnson, C. W.; Donohoe, B. S.; Rorrer, N.; McGeehan, J. E.; Austin, H. P.; Allen, M. D. New modified polyethylene terephthalate -digesting enzyme comprising amino acid mutation of an active site residue, is used to degrade a polymer e.g. polyester, aromatic polymer or semi-aromatic polymer and polyethylene terephthalate. WO2019168811-A1.
(8) Payne, C. M.; Knott, B. C.; Mayes, H. B.; Hansson, H.; Himmel, M. E.; Sandgren, M.; Stahlberg, J.; Beckham, G. T., Fungal Cellulases. Chem. Rev. 2015, 115 (3), 1308-1448. DOI: 10.1021/cr500351c
(9) Taniguchi, I.; Yoshida, S.; Hiraga, K.; Miyamoto, K.; Kimura, Y.; Oda, K., Biodegradation of PET: Current Status and Application Aspects. Acs Catalysis 2019, 9 (5), 4089-4105. DOI: 10.1021/acscatal.8b05171
(10) Tournier, V.; Topham, C. M.; Gilles, A.; David, B.; Folgoas, C.; Moya-Leclair, E.; Kamionka, E.; Desrousseaux, M. L.; Texier, H.; Gavalda, S.; Cot, M.; Guémard, E.; Dalibey, M.; Nomme, J.; Cioci, G.; Barbe, S.; Chateau, M.; André, I.; Duquesne, S.; Marty, A., An engineered PET depolymerase to break down and recycle plastic bottles. Nature 2020, 580 (7802), 216-219. DOI: 10.1038/s41586-020-2149-4