新しい視点を獲得して、受信トレイに直接送信することで、より迅速に作業を進めることができます。

安価で耐久性・適応性に優れたプラスチックの生産は、ここ数十年で爆発的に増え、私たちの生活のあらゆるところに浸透しています。しかし、かつて望ましいとされたこのポリマーには大きな問題があります。 プラスチックは分解に数百年かかると言われており、その天文学的生産量は毎年3億5千万トンを超え、プラスチックによる汚染は世界が直面する最も緊急な環境問題のひとつになっています。
年間1億5千万トンもの驚くべき量のプラスチックが埋立地や自然環境の中へと放出され、800万トン以上が河川から世界の海へと運ばれています。 そのほとんどは分解されず、単に微小粒子に細かくなっていくだけです。 このマイクロプラスチックについては十分に立証されていますが、今や海水中や海洋動物の体内、さらには人間の胃腸の奥深くにまで存在しています。 プラスチック汚染は、現在人類が直面している最も重大な環境問題のひとつであり、研究者はこのややこしいプラスチック問題の解決策を求めて懸命に研究を進めています。
解重合 - ポリマーのリサイクルという問題の解決に向けて
プラスチックは、モノマーブロックが鎖状に繰り返しつながったポリマーでできています。 プラスチックで広く使われているもののほとんどは、熱可塑性または熱硬化性のものです。 アクリル、ポリアミド、ポリエチレンなどの熱可塑性プラスチックは、高温になると柔らかくなり成形可能で、冷えると固まります。 この特性により、柔らかくして新しい製品に再成形できるため、リサイクルは比較的容易ですが、品質は低下するので利点は制限されます。 ポリウレタンやエポキシ樹脂、メラミン樹脂などの熱硬化性プラスチックは、熱を加えると固まるため、リサイクルはほぼ不可能です。 熱可塑性樹脂と熱硬化性樹脂のリサイクルに課題があるということは、すべてのプラスチックがいずれは環境汚染の一因となることを意味しています。
真のリサイクルを実現し、新たな製品に再利用するには、解重合という工程を経て、廃プラスチックを元のモノマーに戻す必要があります。 これは、グローバルな循環型資源経済を実現するために必要な、重要な技術的進歩です。 生態系における微生物による摂取と増殖には、モノマーへの完全な解重合が必要な場合があります。
そこで解重合を実現するため、科学者は自然界に目を向け、プラスチックを分解する微生物の酵素を探しています。 2012年、大阪大学の研究者たちは、世界で最も使用されているプラスチックのひとつ、ポリエチレンテレフタレート(PET、CAS 登録番号25038-59-9、式 (C10H8O4)n)を分解する酵素をコンポストの山から発見しました。
この酵素は、 leaf-branch compost cutinase(LLC)と呼ばれ、PETモノマー間の結合を切断します。ただし、PETの軟化温度65℃に耐えられないため、 この温度で数日間作業すると変性し、産業上の実用性は限定的でした。 解重合は溶けたプラスチックでしか発生しないため、酵素は温度が上がっても安定していることが必要です。
無名の土壌細菌による複動型PET解重合反応
PETは熱可塑性プラスチックで、最も広く使用されているポリエステルのひとつです。 世界のPET生産量は、2014年の4,200万トンから2016年には5,000万トンに増加し、2022年には8,700万トンに達するとされています。
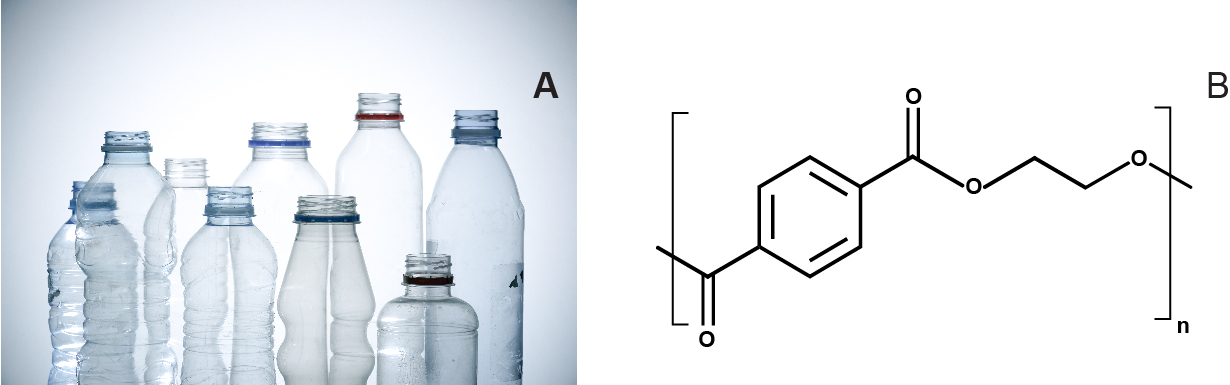
これは石油由来のテレフタル酸(TPA)とエチレングリコール(EG)から製造される合成高分子です。 PETは、結晶構造と粒子径によって、透明、不透明、白色などの色にできる汎用性の高い高分子です(図1)。 衣料用繊維やボトルなどの容器の製造に広く使われており、無延伸PETは熱成形(モールド)によりブリスターパックなど他の梱包容器の製造に使われています1。 PETを効果的に解重合させる方法を発見することは、プラスチックの真のリサイクルとその成果としての環境保護に向けた、重要なマイルストーンになります。
エステラーゼ酵素(エステルを酸とアルコールに分解する酵素)が自然界に多く存在することから、PETの生分解は盛んに研究されてきました2。 しかし、PETの生物学的分解や、微生物の増殖を補助するためのPETの生物学的分解の利用に関する報告はあまりありません。 Fusarium oxysporumやFusarium solaniなど糸状菌のグループの一部の菌は、PETの糸を含む鉱物培地で増殖されています3。
2016年、吉田ら4は、日本のプラスチックリサイクル施設付近のPETに汚染された堆積物で、増殖していた土壌細菌Ideonella sakaiensis 201-F6株を発見し、その特性について報告しました。 このグラム陰性好気性桿菌は、その主要炭素・エネルギー源としてPETを利用して増殖する驚くべき能力をもっています。
このI.sakaiensisは、2つの酵素系を用いてPETをその構成要素のTPAとEGに分解し、それがさらに炭素とエネルギー源に異化します。 2つの酵素の一方のISF6_4831タンパク質は、エステル結合を加水分解して切断します。 脂肪族よりも芳香族のエステルを好み、特にPETを好む傾向があることから、これはPETヒドロラーゼ(PETase)と命名されました。 I. sakaiensis内のPETase酵素はクチナーゼ様のセリンヒドロラーゼで、PETポリマーを攻撃し、ビス(2-ヒドロキシエチル)テレフタラート (BHET)、モノ (2-ヒドロキシエチル) テレフタレート (MHET)、およびTPAを遊離させます。 PETaseは、さらにBHETをMHETとEGに切断します。 第二の酵素のISF6_0224タンパク質、MHETヒドロラーゼ(MHETase)は、可溶性MHETをさらに加水分解してTPAとEGを生成します(図2参照)。 PETを完全にリサイクルするためには、環境に優しいモノマーのTPAとEG4にPETを酵素変換する両方の酵素と、おそらくその相乗効果が必要です。
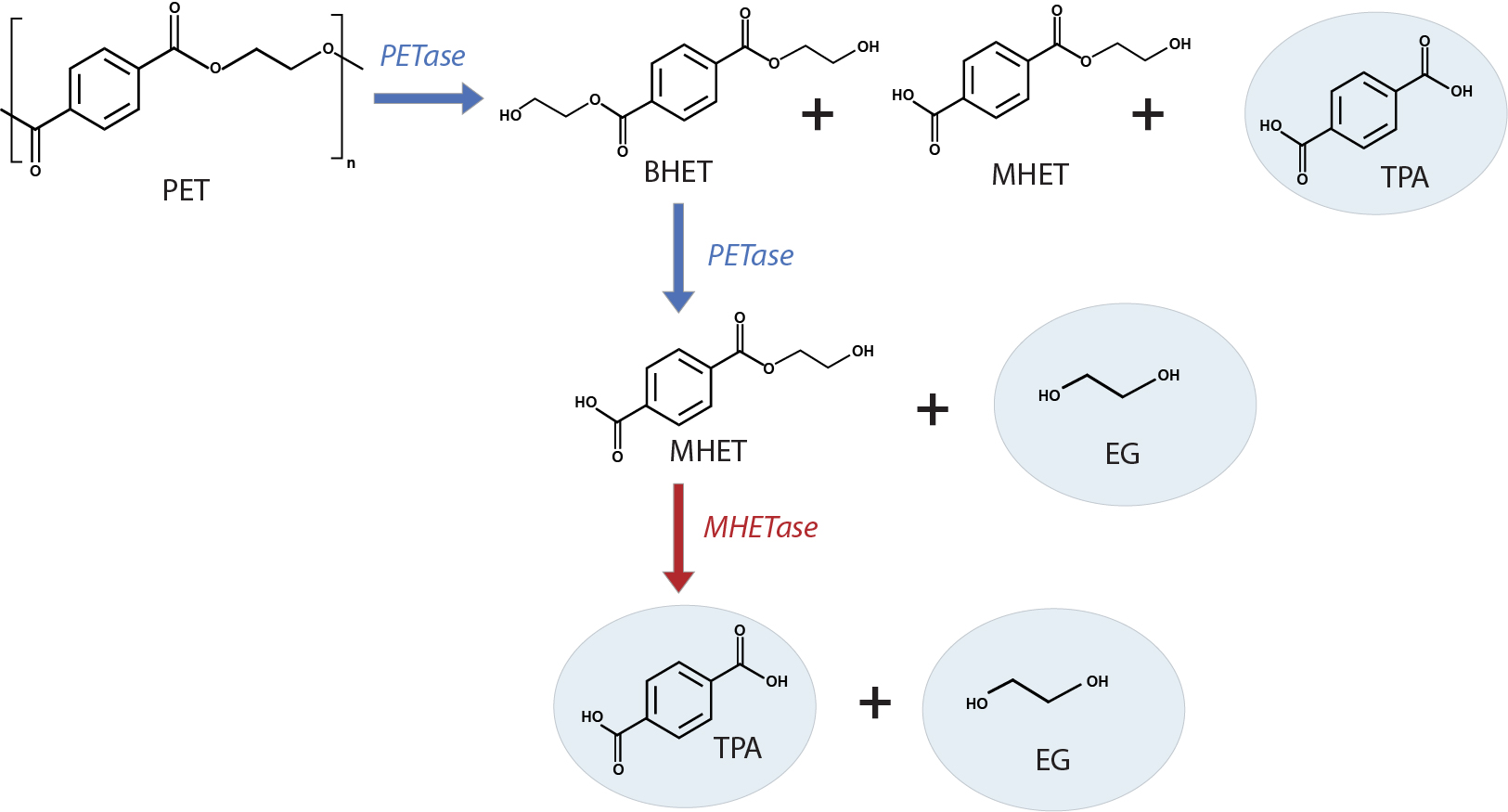
図2. の解重合スキーム:PETaseはその触媒作用でPETをビス(2-ヒドロキシエチル)テレフタラー (BHET)、モノ (2-ヒドロキシエチル) テレフタレート (MHET)とテレフタル酸(TPA)に解重合する。 MHETaseは、MHETをモノマーのTPAとエチレングリコール(EG)に変換する。
PETase変異体によるPET分解能力の大幅向上
PETase酵素の配列と構造の研究では、クチナーゼと類似していることがわかっています。これは、多くの植物において、その保護膜の一部を構成する天然のワックス状の高分子、クチンを分解するために多くの細菌が進化させてきた酵素です。 I.sakaiensis 2のPETaseは、結晶構造解析と生化学的試験により、結合部位にオープンな活性部位構造を持ち、標準的なセリンヒドロラーゼの触媒機構に沿って作動している可能性が高いことが明らかになりました5。
PETaseの構造変化と同族のクチナーゼ活性部位溝に基づき、現在は触媒中心から遠位に二重変異を持つ変異体など、PETaseの変異体が作られ、そしてPET分解の試験が行われています。 この領域は、重要な基質結合相互作用を改良できると仮定されています。 このクチナーゼ構造に基づく二重変異体は、野生型PETaseに比べて高いPET分解能力を示すことがわかり6、現在特許を申請中です7。
クチナーゼの2つの活性部位の残基の変異によって結合溝を狭めることで、PETの分解が促進されることが確認されています。これは、PETが豊富な環境で進化したにもかかわらず、PETaseが結晶性PETの分解に最適な構造を示していないことを示唆するものです。 この変異型の酵素では、わずか数日後からプラスチックの分解が始まります。海洋での分解に数百年かかるのに比べると格段に速いと言えます。
二重変異体から二重酵素カクテルへ
この反応にMHETase酵素を加えると、混合酵素はPETase単体よりも2倍速くPETを分解します。 テストした酵素の負荷量範囲内で観察された分解の傾向は、両酵素の濃度が高くなるにつれて構成モノマーレベルが上昇することを示しています。 これは、反応が基質ではなく酵素に制限されていることを示します。 また、相乗効果分析により、PETaseの負荷により分解率が上昇すること、PETaseに対して低濃度であってもMHETaseが存在することで総分解率が向上することが示されました。 今回の実験では、PETaseとMHETaseの最適な比率は示されていません。
スーパー酵素作製でPET分解が3倍に
PETの分解特性とその範囲を探る追加の実験の中で、研究者たちは、MHETaseとPETaseを1本の長い鎖につなぎ合わせて新たなスーパー酵素を生み出しました。 柔軟なグリシン・セリンリンカーを用いて、MHETaseのC末端とPETaseのN末端を共有結合したキメラタンパク質を生成し、非晶質PETの分解を分析試験しました(図3)。 異なる複数の酵素によって達成された分解の程度を比較すると、キメラタンパク質はPETaseとMHETaseの両方、および非連結の酵素混合物よりも優れていることがわかりました。
興味深いことに、このスーパー酵素はPETを解重合するだけでなく、ビール瓶に使われている糖由来のバイオプラスチックであるPEF(ポリエチレンフラノエート)にも作用します。
セルロースやキチンなどの天然高分子の酵素分解は、自然界では微生物から分泌される相乗作用する酵素の混合物によって達成されています8。 これらの天然微生物のシステムは、こういった高分子を最適に分解できるよう、長い時間をかけて進化してきました。 I. sakaiensisなどの一部の土壌細菌も、同様に2つの酵素系でポリエステル基質を利用するために進化してきたようです4,9。 何世紀もかかる自然分解とは異なり、スーパー酵素はわずか数日でPETをモノマーに戻せます。とは言え、まだこれでは遅すぎて商業的に採算が合いません。
プラスチックの分解で無限のリサイクル
スーパー酵素は、PETを元のモノマーに戻すことで、プラスチックを無限に製造そして再利用することを可能にし、化石資源への依存を減らすことができます。 しかも技術革新はそこで終わりません。
2020年、PETをわずか10時間で分解する別の酵素が発見されるという、大きな進展がありました10。 研究では、2012年に初めて発見された葉枝コンポストのクチナーゼ(LCC)をはじめ、多種多様な菌や酵素をスクリーニングし、候補の酵素を探しました。そして、結合部位のアミノ酸を変え、熱安定性を向上させることで、数百種類の変異型PETヒドロラーゼ酵素を作製しました。 その後、細菌変異体をスクリーニングし、効率的なPET分解酵素を同定しました。 この作業を何度も繰り返し、PETを分解する効率が元のLCCの1万倍も高い変異型酵素を単離したのです。 これは、PETの融点に近い72℃でも安定しています。 この発見は、PETの無限リサイクルの実現に大きく貢献するものです。実際に、すでに工業における試運転段階に入っています10。
微生物とその酵素がもたらす可能性は、まだ氷山の一角に過ぎません。 ほとんどのプラスチックは化石燃料から作られており、よってその製造は有限ですが、にもかかわらず私たちの環境にはどこにでもあります。 循環経済を形成する方法を見出さない限り、プラスチック汚染は今後も拡大し続けるでしょう。 すでに存在する廃棄物をリサイクルする方法を見つけなければ、私たちが依存しているプラスチック製品は数十年後には生産できなくなります。 従来のリサイクルは効果的でも持続可能でもなく、工業規模でプラスチックをモノマー成分に還元しない限り、この問題の解決は望めません。 幸い、自然の助けと技術を尽くした進化、そして科学的な工夫によって、この問題は解決できる希望が出てきました。
「グリーン」なイノベーションをさらに知る
イノベーションの原動力になる新種を探す上で、ブラジルの熱帯雨林ほど生物多様性が豊かな場所はありません。 データがどのようにして収集され、公的に利用されているか、そしてそうすることでそれが地球上の生物多様性の15~20%を保護することにどう結びつくのか、より詳しく知りましょう。
(『重要なデータの構築によりブラジルの生物多様性からイノベーション実現へ』をお読みください)
参考文献
(1) Pasbrig, E.; Claessens, P.; Walker, R. I.; Walker, R. Peelable cover film for pharmaceutical packaging, e.g. blister packs, comprises paper, aluminum foil or heat-resistant plastic, a layer of special plastic film, mesh or fabric, a layer of aluminum foil and a heat-sealing layer. EP1767347-A1; WO2007038488-A2; EP1928654-A2; AU2006294788-A1; US2008251411-A1; CN101316702-A; CA2623586-A1; JP2009509874-W; TW200727887-A; MX2008004201-A1; IN200801248-P2; ZA200802826-A; BR200616412-A2; WO2007038488-A3; EP1928654-A4.
(2) Han, X.; Liu, W. D.; Huang, J. W.; Ma, J. T.; Zheng, Y. Y.; Ko, T. P.; Xu, L. M.; Cheng, Y. S.; Chen, C. C.; Guo, R. T., Structural insight into catalytic mechanism of PET hydrolase. Nature Communications 2017, 8. DOI: 10.1038/s41467-017-02255-z
(3) Nimchua, T.; Eveleigh, D. E.; Sangwatanaroj, U.; Punnapayak, H., Screening of tropical fungi producing polyethylene terephthalate-hydrolyzing enzyme for fabric modification. J. Ind. Microbiol. Biotechnol. 2008, 35 (8), 843-850. DOI: 10.1007/s10295-008-0356-3
(4) Yoshida, S.; Hiraga, K.; Takehana, T.; Taniguchi, I.; Yamaji, H.; Maeda, Y.; Toyohara, K.; Miyamoto, K.; Kimura, Y.; Oda, K., A bacterium that degrades and assimilates poly(ethylene terephthalate). Science 2016, 351 (6278), 1196-1199. DOI: 10.1126/science.aad6359
(5) Rauwerdink, A.; Kazlauskas, R. J., How the Same Core Catalytic Machinery Catalyzes 17 Different Reactions: the Serine-Histidine-Aspartate Catalytic Triad of alpha/beta-Hydrolase Fold Enzymes. Acs Catalysis 2015, 5 (10), 6153-6176. DOI: 10.1021/acscatal.5b01539
(6) Austin, H. P.; Allen, M. D.; Donohoe, B. S.; Rorrer, N. A.; Kearns, F. L.; Silveira, R. L.; Pollard, B. C.; Dominick, G.; Duman, R.; El Omari, K.; Mykhaylyk, V.; Wagner, A.; Michener, W. E.; Amore, A.; Skaf, M. S.; Crowley, M. F.; Thorne, A. W.; Johnson, C. W.; Woodcock, H. L.; McGeehan, J. E.; Beckham, G. T., Characterization and engineering of a plastic-degrading aromatic polyesterase. Proc. Natl. Acad. Sci. U. S. A. 2018, 115 (19), E4350-E4357. DOI: 10.1073/pnas.1718804115
(7) Beckham, G. T.; Johnson, C. W.; Donohoe, B. S.; Rorrer, N.; McGeehan, J. E.; Austin, H. P.; Allen, M. D. New modified polyethylene terephthalate -digesting enzyme comprising amino acid mutation of an active site residue, is used to degrade a polymer e.g. polyester, aromatic polymer or semi-aromatic polymer and polyethylene terephthalate. WO2019168811-A1.
(8) Payne, C. M.; Knott, B. C.; Mayes, H. B.; Hansson, H.; Himmel, M. E.; Sandgren, M.; Stahlberg, J.; Beckham, G. T., Fungal Cellulases. Chem. Rev. 2015, 115 (3), 1308-1448. DOI: 10.1021/cr500351c
(9) Taniguchi, I.; Yoshida, S.; Hiraga, K.; Miyamoto, K.; Kimura, Y.; Oda, K., Biodegradation of PET: Current Status and Application Aspects. Acs Catalysis 2019, 9 (5), 4089-4105. DOI: 10.1021/acscatal.8b05171
(10) Tournier, V.; Topham, C. M.; Gilles, A.; David, B.; Folgoas, C.; Moya-Leclair, E.; Kamionka, E.; Desrousseaux, M. L.; Texier, H.; Gavalda, S.; Cot, M.; Guémard, E.; Dalibey, M.; Nomme, J.; Cioci, G.; Barbe, S.; Chateau, M.; André, I.; Duquesne, S.; Marty, A., An engineered PET depolymerase to break down and recycle plastic bottles. Nature 2020, 580 (7802), 216-219. DOI: 10.1038/s41586-020-2149-4